La conservation de l’information génétique au cours de sa réplication
-
I)Le mécanisme de la conservation de l’information génétique au cours de la réplication
-
-
1)le doublement de la quantité d’ADN précède toute division de la cellule
-
-
-
-
•Il existe un processus qui permet de doubler la quantité d’ADN et de passer de 2n chromosome avec 1 chromatide à 2n chromosomes avec 2 chromatides.
-
-
2)Une réplication semi-conservative et semi-discontinue
-
-
A)mise en évidence du caractère semi-conservatif de la réplication
-
-
-
•La réplication semi-conservative a été mise en évidence par Mesglson et Stahl (1958) , et 10 ans après Okasaki ( 1968 ) montre que cette réplication est, en plus d’être semi-conservative, semi-discontinue.
-
•Mesglson et Sthal nourrissent des colibacilles avec de l’azote lourd, qui est intégré dans l’ADN. Après centrifugation avec du chlorure de césium, ils obtiennent des bandes de densité 1,8 . Ils cultivent ensuite les bactéries sur de l’azote léger, et après une même centrifugation, ils obtiennent un bande de densité 1,72. À la troisième génération ils obtiennent 2 bandes de densité 1,72 et 1,65.
-
•Cairns à confirmer cette théorie par des expériences de marquages. Après marquage à la thymidine tritiée et radiographie, il obtient ceci :

Il observe une zone plus dense aux électrons qu’il interprète comme une zone avec 2 brins marqués. Cette «oeil» est un témoin d’une réplication à 2 sens.
-
•Dans un deuxieme temps, il observe :



-
•Chez les eucaryotes, l’observation de nucléofilaments permet de voir des yeux de réplications très nombreux. On trouve 500 points d’origine de réplication chez la levure, près de 100 000 ORI chez l’homme.
-
-
-
B)Conditions de la réplication
-
-
-
-
-
•Kornberg a montré que l’ADN synthétisé est identique à l’ADN de départ et que la chaîne polynucléotidique néosynthétisée croît dans le sens 5’ ➞ 3’ . La réplication repose sur la complémentarité des bases.
-
•La synthèse d’ADN nécessite une enzyme (ADN polymérase), une amorce d’ADN préformé, du Mg2+ et de l’ATP.
-
-
-
C)Une réplication semi-discontinue
-
-
-
-
•Okasaki montre que la réplication n’est pas que semi conservative du fait que l’ADN polymérase ne fonctionne que dans un sens.
-
•Il y a sur l’un des deux brins une synthèse continue (brin précoce), et sur l’autre brin il y a des fragments qui sont synthétisés (fragments d’Okasaki).
-
•Ce phénomène mis en évidence par Okasaki est dû à l’activité dans le sens 5’ ➞ 3’ de l’ADN polymérase.
-
-
3)Mécanismes moléculaires de la réplication
-
-
-
-
A)La réplication est bidirectionnelle
-
-

-
•La réplication est bidirectionnelle : dans les 2 sens chez les procaryote.

Etape de synthèse de l’ADN sur le brin tardif:
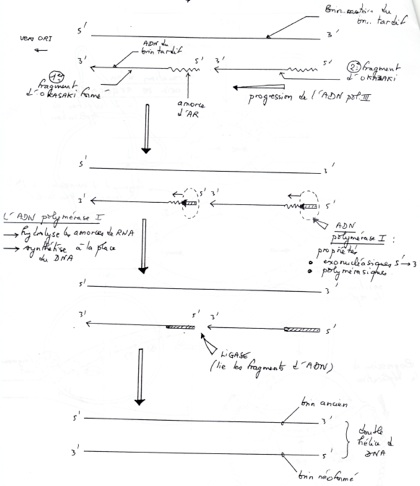
-
•Dans la cellule :
-
•L’ADN polymérase I digère les morceaux d’ARN et le remplace par de l’ADN. A l’issue de ce travail, tout les fragment d’Okasaki sont de l’ADN
-
•Puis une ligase permet de relier entre eux les fragment d’ADN, qui forment donc ainsi un brin continu.
-
-
-
-
B)Les polymérases et leur mécanisme d’action
-
-
-
-
•Diversité et propriétés des ADN polymérases: chez les procaryotes, 3 ADN polymérases ont été caractérisées dont :
-
•ADN polymérase III : assure l’élongation de l’ADN au cours de la réplication.
-
•Fidélité : grâce à son activité exonucléasique 3’ ➞ 5’, c’est à dire qu’elle est capable de corriger ces erreur de transcription.
-
•Processivité : catalyse de nombreuses réactions sans libérer son substrat. L’ADN reste fixé à l’enzyme.
-
•Potentiel catalytique : 1000 nucléotides/s
-
•ADN polymérase I ( =ADN polymérase de Kornberg) qui réalise simultanément :
-
•La dégradation des amorces d’ARN = activité exonucléasique 5’ ➞ 3’
-
•Leur remplacement par de l’ADN = activité polymérasique 5’ ➞ 3’
-
•ADN polymérase II : participe aux mécanismes de réparation de l’ADN.
-
Chez les eucaryotes, on connaît au moins 13 polymérases :
Polymérase ε : synthèse du brin continu
Polymérase δ : synthèse du brin discontinu
Polymérase α : primase
Polymérase β : réparation
Polymérase γ : synthèse et réparation de l’ADN mitochondriale.
-
•Organisation structurale et mécanisme d’action :
-
•ADN polymérase d’E.coli
-
•
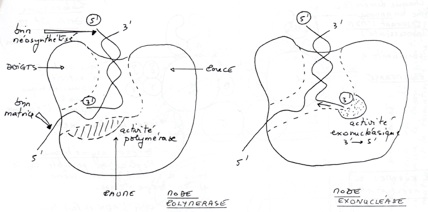
-
•3 domaines structuraux = 3 domaines fonctionnelles :
-

-
•ADN polymérase III d’E.Coli
-

Dimère asymétrique qui peut être mis en lien avec la réplication asymétrique. Cette enzyme est entièrement protéique.
-
•Mécanisme d’action.
-
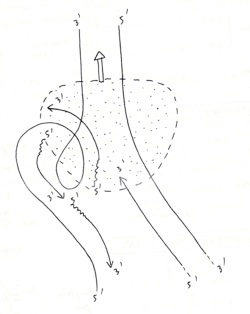
•Ouverture de la double hélice et stabilisation des brins:
-
•Hélicase + ATP : proteine initiatrices : dissocie les paires de bases. Rompt les liaison hydrogènes sur 1 tour d’hélice = 10 paires de bases ➞ ouverture de la double hélice.
-
•Topoisomérases II (= gyrase) + ATP : produit un sur-enroulement négatif, et supprime les contraintes de torsions qui s’accumulent du fait de l’ouverture de l’hélice.
-
•Protéine SSB (Single Strand DNA Binding)
-
•Synthèse de nouveaux brins d’ADN
-
•ADN polymérase III
-
•ARN polymérase = primase : synthèse d’amorces d’ARN de 10 nucléotides environ, qui est un préalable de la synthèse des fragments d’Okasaki.
-
•ADN polymérase I
-
•ADN Ligase : soude les fragments d’Okasaki après que l’ADN polymérase I est remplacer l’ADN par de l’ARN.
-


Les protéines SSB : empêchent réappariemment des brins, empêchent un brin de se replier sur lui même.

-
II)Fidélité de la copie qui n’est que partielle
-
-
1)Vulnérabilité de l’ADN et glissement des polymérases
-
-
-
•Sites vulnérables chimiquement :


-
-
•Il peut aussi y avoir un hydrolyse avec rupture du squelette Ose-P
-
•Il peut y avoir oxydation des bases ou du désoxyribose
-
•Il peut y avoir rupture du cycle.
-
-
-
•Insertions et délétions par glissement et décalage de la polymérase.
-
•Exemple d’une addition : phénomène fréquent dans région d’ADN avec des séquences répétées (= «points chauds» mutationnels )
-
-
2)Des mutations variées peuvent modifier le message génétique
-
-
-
A)Définition et caractère des mutations
-
-
-
•Les mutations sont des modifications touchant l’ADN. Ces mutations peuvent être ponctuelles, ou par segments, ou encore sur des chromosomes entiers. Les causes de ces mutations peuvent être environnementales, ou réplicationnelles ou autres. Ces mutations peuvent se produire à n’importe quel moment du cycle cellulaire.
-
•Caractéristique d’une mutation :
-
•Rareté : taux de mutation ou fréquence de mutations est le nombre de mutation par locus et par génération cellulaire. Ce taux est de 10-6 à 10-11. En moyenne, pour le gène Lac chez E.Coli est de 10-7.
-
•Aléatoire : la nature de la mutation ne dépend pas des conditions externes, même si la fréquence peut être augmentée par des facteurs externes. On peut provoquer des mutations, on sait même diriger expérimentalement les mutations (qui ne sont donc plus aléatoire)
-
•La mutation peut être soit germinale, soit somatique
-
•Réversibilité très rare : 10-7 x 10-7 = 10-14
-
•La plupart des mutation géniques sont létales (90%)
-
•Existence de «points chauds» mutationnels (microsatellites)
-
-
•Taux ou fréquence de mutation = μ = probabilité de mutation par locus et par génération cellulaire. A ne pas confondre avec la fréquence de mutants, fréquence à laquelle on trouve une mutation donnée dans un population de cellule ou d’individus. Peut être beaucoup plus élevé que le taux de mutation. (Si une mutation a eu lieu au cycle cellulaire antérieur à celui ou on a fait le dénombrement mutée du cycle antérieur se sera multiplié au cycle suivant)
-
-
-
B)La tautomérisation : une cause d’erreurs lors de la réplication (fréquence : 10-4 à 10-5)
-
-
-
-
•Analyse des conséquences de la tautomérie :


-
-
-
C)Diversité des mutation par substitution d’une paire de bases
-
-
-
-
•L’ADN est vulnérable et peut subir des altérations qui ont pour conséquences des mutations après la réplication. Mais il existe des mécanismes qui régulent ces mutations, et réparent ces altérations.
-
•Mutation par transition : 4 possibles. Remplacement d’une base d’un type par une base de même type
-
•Mutation par transversion : 8 possibles. Remplacement d’une base purique par une base pyrimidique ou réciproquement.
-
-
III)Mécanismes assurant la fidélité de la réplication
-
-
1)Une spécificité élevée de l’ADN polymérase III
-
-
-
•Stabilité du complexe formé par :
-
•ADN polymérase III
-
•Le brin d’ADN matrice
-
•Le NTP
-
•La stabilité est maximale lorsque le NTP est complémentaire du nucléotide de la matrice. Erreur de l’ordre de 1 nucléotide non complémentaire sur 105.
-
-
2)Utilisation de la thymine à la place de l’Uracile dans l’ADN
-
-
-
•Lors de la désamination d’une cytosine, elle devient une uracile. Si la thymine n'était pas utilisée dans l’ADN, l’uracile correctement en place serait indistinguable de l’uracile issue de la désamination de la cytosine, ce qui enlève toute possibilité de réparation d’une mutation.
-
-
3)Une correction sur épreuve au cours de la réplication
-
-
-
•Une des conséquences des mutations est la mise en oeuvre de mécanismes correcteurs des mutations. C’est l’activité exonucléasique 3’➞5’ de la polymérase III. Cela permet de remplacer un nucléotide mal apparié.
